
En skärmdump av DIVE -webbplatsen. Kredit:Gupta et al.
Akademiska uppsatser innehåller ofta redogörelser för nya genombrott och intressanta teorier relaterade till en mängd olika områden. Dock, de flesta av dessa artiklar är skrivna med jargong och tekniskt språk som bara kan förstås av läsare som är bekanta med just det ämnesområdet.
Icke-expertläsare kan därför vanligtvis inte förstå vetenskapliga artiklar, om de inte samlas in och görs mer tillgängliga av tredje part som förstår begreppen och idéerna som finns i dem. Med detta i åtanke, ett team av forskare vid Texas Advanced Computing Center vid University of Texas i Austin (TACC), Oregon State University (OSU) och American Society of Plant Biologists (ASPB) har bestämt sig för att utveckla ett verktyg som automatiskt kan extrahera viktiga fraser och terminologi från forskningsartiklar för att ge användbara definitioner och förbättra deras läsbarhet.
"Vårt projekt motiveras av behovet av att förbättra läsbarheten för tidningsartiklar, "Weijia Xu, som leder teamet på TACC, berättade TechXplore. "Det är ett gemensamt arbete mellan biologiska kuratorer, tidskriftsutgivare och datavetenskapare som syftar till att utveckla en webbtjänst som kan känna igen och möjliggöra författarens kurering av viktig terminologi som används i tidskriftspublikationer. Terminologin och orden bifogas sedan i slutet av tidningsartikeln för att öka dess tillgänglighet för läsare. "
Xu och hans kollegor utvecklade ett utbyggbart ramverk som kan användas för att extrahera information från dokument. De implementerade sedan detta ramverk inom en webbtjänst som heter DIVE (Domain Information Vocabulary Extraction), integrera den med tidskriftens publikationspipeline för ASPB. Till skillnad från befintliga verktyg för att extrahera domäninformation, deras ramverk kombinerar flera tillvägagångssätt, inklusive ontologi-styrd extraktion, regelbaserad extraktion, naturlig språkbehandling (NLP) och djupinlärningsteknik.

Arkitekturöversikten över systemet som forskarna föreslog. Kredit:Gupta et al.
"Resultaten från olika modeller lagras sedan i en centraliserad databas, "Xu förklarade." Vi har också utformat en webbtjänst som gör det möjligt för användare att ta fram extraktionsresultat. Webbtjänsten är integrerad med produktionspublikationsrörledningen på ASPB. "
När förhandsversionen av en tidningsartikel har skickats in och kommer in i ASPB:s pipeline, manuskriptet matas automatiskt till DIVE, som behandlar den och producerar en webbadress som författaren kommer att kunna komma åt bearbetningsresultaten för DIVE. Författaren till uppsatsen uppmanas att besöka länken som tillhandahålls och granska den extraherade informationen innan han/hon officiellt kan lämna in papperet.
"Författaren måste besöka DIVE -sajten för att granska extraktionsresultaten och slutgiltigt godkänna listan med information som ska ingå i slutet av artikeln, "Sa Xu." DIVE spårar också författarkorrigeringar för att förbättra framtida extraktionsuppgifter. För närvarande, ingen annan tidskriftsutgivare har antagit ett liknande tillvägagångssätt och integrerat det med deras publiceringspipeline. "
Under sina analyser och vid extrahering av nyckeldata från dokument, den ram som forskarna utvecklat använder flera tekniker. Detta gör att den kan fånga mer information än andra metoder, t.ex. ABNER (A Biomedical Named Entity Recognizer), som är ett programvara för öppen källkod för molekylärbiologisk textbrytning som bara kan extrahera allmänna termer (t.ex. gener och proteiner). I motsats till DIVE, ABNER är endast baserat på villkorliga slumpmässiga fält (CRF), en statistisk modelleringsmetod som vanligen används i mönsterigenkänning och applikationer för maskininlärning.
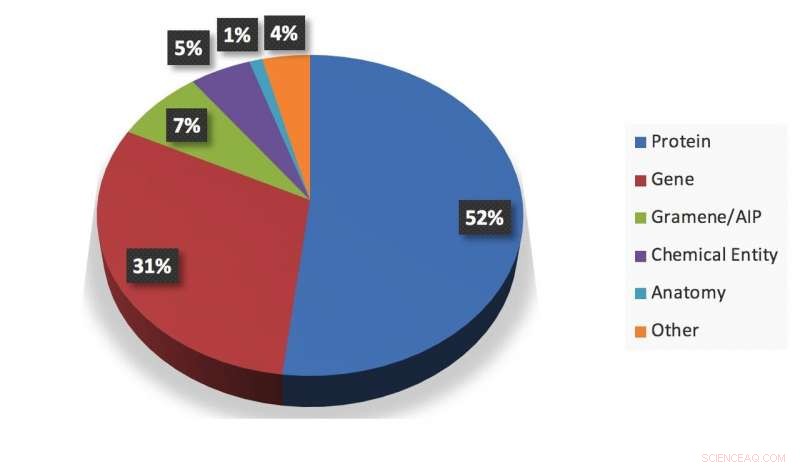
En visuell sammanfattning av en ögonblicksbild av information som extraherats av systemet. Kredit:Gupta et al.
"Ett stort bidrag i vårt projekt är att det hjälper till att bygga datamängder och modeller som kan härleda författares forskningsintressen från deras publikationer, "Xu sa." Vårt projekt kan gynna bredare grupper av biologiska forskare. För författare, extraktionen och införandet av nyckelinformationen kan öka tillgängligheten för deras artiklar. "
Xu och hans kollega Amit Gupta utvärderade sin ram och jämförde dess prestanda med andra informationsutvinningverktyg, inklusive ABNER. Deras resultat avslöjade att med hjälp av flera metoder, inklusive djupinlärning, DIVE uppnår högre precision än andra förutbildade modeller enbart baserade på CRF. Intressant, DIVE -ramverket kan också uppdateras kontinuerligt, eftersom ytterligare extraktionsmodeller kan läggas till den när som helst.
DIVE-webbapplikationen tillåter inte bara icke-expertläsare att bättre förstå akademiska uppsatser, det kan också hjälpa dem att identifiera papper i linje med deras intressen. Forskare, å andra sidan, kan använda DIVE för att hålla sig informerad om specifika forskningsområden, samt att lära sig om ny terminologi och trender relaterade till deras intresseområde. Till sist, informationen som genereras av applikationen kan också vägleda biologikuratorer i deras beslut och datainsamlingsprocesser.
"Vi fortsätter vårt projekt genom att utforska två riktningar, "Sa Xu." Å ena sidan, vi undersöker nya metoder att integrera med våra informationsutvinning modeller för att förbättra prestanda. Å andra sidan, Vi försöker också utöka vår tjänst genom att erbjuda den till fler användargrupper och tidskriftsutgivare. "
© 2019 Science X Network