Genomsekvenseringsteknik ger tusentals nya växtgenom årligen. Inom jordbruket slår forskare samman denna genomiska information med observationsdata (som mäter olika växtegenskaper) för att identifiera korrelationer mellan genetiska varianter och grödans egenskaper som fröantal, resistens mot svampinfektioner, fruktfärg eller smak.
Uppfattningen om hur genetisk variation påverkar genaktivitet på molekylär nivå är dock ganska begränsad. Denna kunskapslucka hindrar förädling av "smarta grödor" med förbättrad kvalitet och minskad negativ miljöpåverkan som uppnås genom kombination av specifika genvarianter med känd funktion.
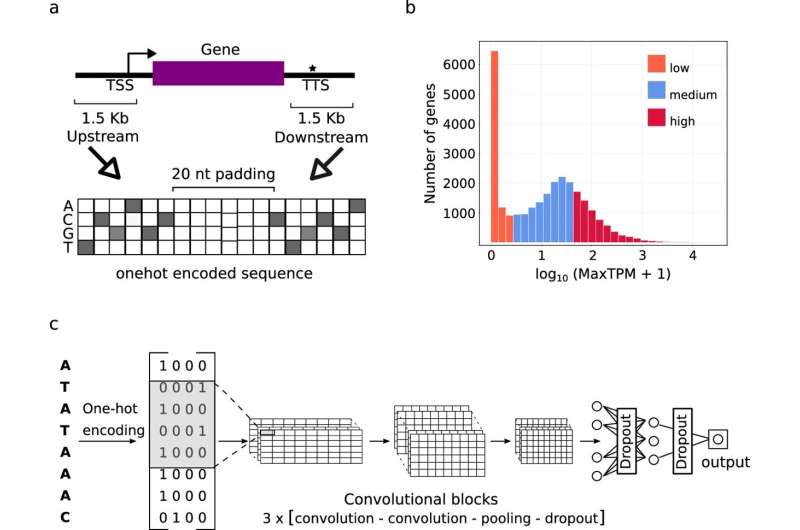
Forskare från IPK Leibniz Institute och Forschungszentrum Jülich (FZ) har gjort ett betydande genombrott för att tackla denna utmaning. Under ledning av Dr. Jedrzej Jakub Szymanski tränade det internationella forskarteamet tolkbara modeller för djupinlärning, en delmängd av AI-algoritmer, på ett stort dataset av genomisk information från olika växtarter.
"Dessa modeller kunde inte bara exakt förutsäga genaktivitet från sekvenser utan också fastställa vilka sekvensdelar som bidrar till dessa förutsägelser", förklarar chefen för IPK:s forskargrupp "Nätverksanalys och modellering." AI-tekniken som forskarna tillämpade liknar den som används i datorseende, vilket innebär att känna igen ansiktsdrag i bilder och härleda känslor.
I motsats till tidigare tillvägagångssätt baserade på statistisk anrikning, kombinerade forskarna här identifiering av sekvensegenskaper med bestämning av mRNA-kopiantalet inom ramen för en matematisk modell som har tränats redogöra för biologisk information om genmodellstruktur och sekvenshomologi, alltså gen evolution.
"Vi blev verkligen förvånade över effektiviteten. Inom några dagars träning återupptäckte vi många kända regulatoriska sekvenser och fann att cirka 50 % av de identifierade funktionerna var helt nya. Dessa modeller generaliserade utmärkt över växtarter som de inte tränats på, vilket gör dem värdefulla för att analysera nyligen sekvenserade genom", säger Dr. Szymanski.
"Och vi demonstrerade specifikt deras tillämpning i olika tomatsorter med långlästa sekvenseringsdata. Vi hittade specifika regulatoriska sekvensvariationer som förklarade observerade skillnader i genaktivitet och följaktligen variationer i form, färg och robusthet. Detta är en anmärkningsvärd förbättring jämfört med klassiskt använda statistiska associationer av enkelnukleotidpolymorfismer."
Teamet har öppet delat med sig av sina modeller och tillhandahållit ett webbgränssnitt för deras användning. "Intressant nog, mycket ansträngning lades ner på att försämra vår modells prestanda. För att undvika alltför optimistiska resultat på grund av att AI hittade genvägar krävdes en djupdykning i genregleringsbiologin för att eliminera eventuella fördomar, minska dataläckage och överanpassning", säger Fritz Forbang Peleke, ledande forskare inom maskininlärning och första författare till studien, som publicerades i tidskriften Nature Communications .
Dr Simon Zumkeller, en medförfattare och evolutionsbiolog från FZ Jülich, säger:"Med de presenterade analyserna kan vi undersöka och jämföra genreglering i växter och sluta sig till dess utveckling. För praktiska tillämpningar ger metoden också en ny grund. Vi närmar oss rutinmässig identifiering av genreglerande element i kända och nysekvenserade växtgenom, i olika vävnader och under olika miljöförhållanden."
Mer information: Fritz Forbang Peleke et al, Deep learning the cis-regulatory code for genexpression in valda modellväxter, Nature Communications (2024). DOI:10.1038/s41467-024-47744-0
Journalinformation: Nature Communications
Tillhandahålls av Leibniz Institute of Plant Genetics and Crop Plant Research