Att observera enskilda celler genom mikroskop kan avslöja en rad viktiga cellbiologiska fenomen som ofta spelar en roll i mänskliga sjukdomar, men processen att särskilja enskilda celler från varandra och deras bakgrund är extremt tidskrävande – och en uppgift som är väl lämpad för AI-hjälp.
AI-modeller lär sig hur man utför sådana uppgifter genom att använda en uppsättning data som kommenteras av människor, men processen att särskilja celler från deras bakgrund, kallad "encellssegmentering", är både tidskrävande och mödosam. Som ett resultat finns det en begränsad mängd kommenterad data att använda i AI-träningsset. Forskare från UC Santa Cruz har utvecklat en metod för att lösa detta genom att bygga en AI-modell för generering av mikroskopbilder för att skapa realistiska bilder av enstaka celler, som sedan används som "syntetisk data" för att träna en AI-modell för att bättre kunna utföra encellssegmentering.
Den nya programvaran beskrivs i en ny artikel publicerad i tidskriften iScience . Projektet leddes av biträdande professor i biomolekylär teknik Ali Shariati och hans doktorand Abolfazl Zargari. Modellen, kallad cGAN-Seg, är fritt tillgänglig på GitHub.
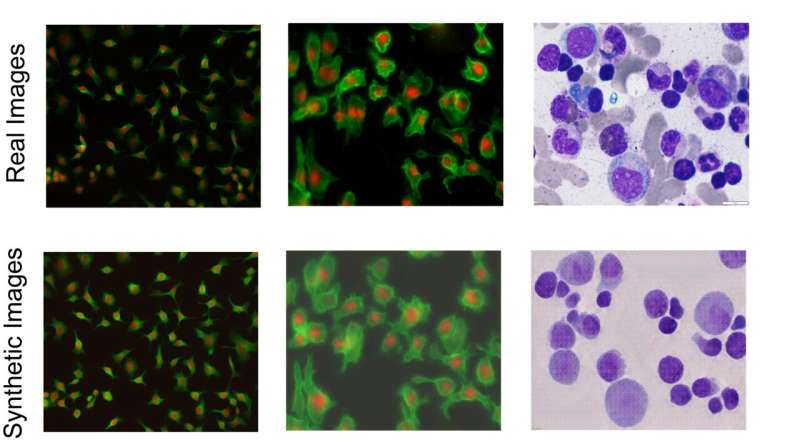
"Bilderna som kommer ut ur vår modell är redo att användas för att träna segmenteringsmodeller," sa Shariati. "På sätt och vis gör vi mikroskopi utan mikroskop, genom att vi kan generera bilder som är mycket nära verkliga bilder av celler när det gäller de morfologiska detaljerna i den enskilda cellen. Det fina med det är att när de kommer ut av modellen är de redan kommenterade och märkta. Bilderna visar massor av likheter med riktiga bilder, vilket gör att vi kan skapa nya scenarier som inte har setts av vår modell under utbildningen."
Bilder av enskilda celler sedda genom ett mikroskop kan hjälpa forskare att lära sig om cellbeteende och dynamik över tid, förbättra sjukdomsupptäckt och hitta nya läkemedel. Subcellulära detaljer som textur kan hjälpa forskare att svara på viktiga frågor, som om en cell är cancerös eller inte.
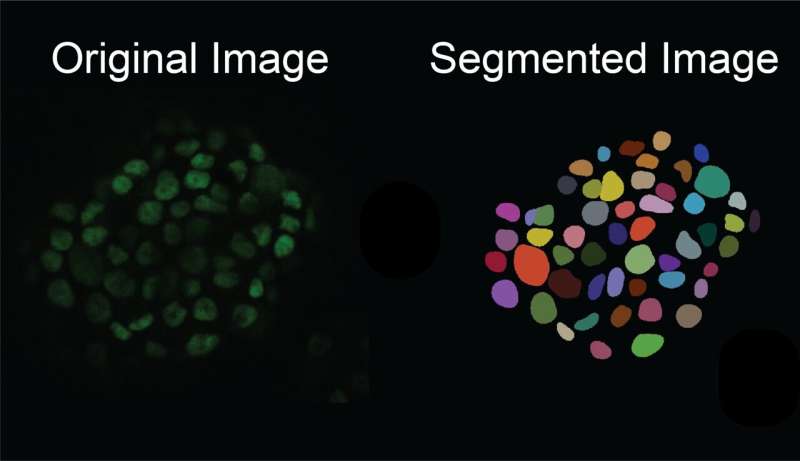
Att manuellt hitta och märka cellers gränser utifrån deras bakgrund är dock extremt svårt, särskilt i vävnadsprover där det finns många celler i en bild. Det kan ta forskare flera dagar att manuellt utföra cellsegmentering på bara 100 mikroskopibilder.
Djup inlärning kan påskynda denna process, men en första datamängd med kommenterade bilder behövs för att träna modellerna – åtminstone tusentals bilder behövs som baslinje för att träna en korrekt modell för djupinlärning. Även om forskarna kan hitta och kommentera 1 000 bilder, kanske dessa bilder inte innehåller den variation av funktioner som visas under olika experimentella förhållanden.
"Du vill visa att din djupinlärningsmodell fungerar över olika prover med olika celltyper och olika bildkvaliteter," sa Zargari. "Om du till exempel tränar din modell med bilder av hög kvalitet, kommer den inte att kunna segmentera de lågkvalitativa cellbilderna. Vi kan sällan hitta en så bra datamängd i mikroskopifältet."
För att ta itu med detta problem skapade forskarna en bild-till-bild-generativ AI-modell som tar en begränsad uppsättning kommenterade, märkta cellbilder och genererar fler, som introducerar mer intrikata och varierande subcellulära funktioner och strukturer för att skapa en mångsidig uppsättning "syntetiska" bilder. Särskilt kan de generera kommenterade bilder med en hög täthet av celler, som är särskilt svåra att kommentera för hand och är särskilt relevanta för att studera vävnader. Den här tekniken fungerar för att bearbeta och generera bilder av olika celltyper såväl som olika avbildningsmodaliteter, till exempel de som tagits med fluorescens eller histologisk färgning.
Zargari, som ledde utvecklingen av den generativa modellen, använde en allmänt använd AI-algoritm som kallas ett "cykelgenerativt motståndsnätverk" för att skapa realistiska bilder. Den generativa modellen är förstärkt med så kallade "augmentation functions" och ett "style injecting network", som hjälper generatorn att skapa en mängd olika högkvalitativa syntetiska bilder som visar olika möjligheter för hur cellerna skulle kunna se ut. Såvitt forskarna vet är detta första gången stilinjiceringstekniker används i detta sammanhang.
Sedan används denna mångsidiga uppsättning syntetiska bilder som skapats av generatorn för att träna en modell för att exakt utföra cellsegmentering på nya, riktiga bilder tagna under experiment.
"Genom att använda en begränsad datamängd kan vi träna en bra generativ modell. Med den generativa modellen kan vi generera en mer mångsidig och större uppsättning av kommenterade, syntetiska bilder. Genom att använda de genererade syntetiska bilderna kan vi träna en bra segmenteringsmodell— det är huvudtanken", sa Zagari.
Forskarna jämförde resultaten av sin modell med hjälp av syntetisk träningsdata med mer traditionella metoder för att träna AI för att utföra cellsegmentering över olika typer av celler. De fann att deras modell ger avsevärt förbättrad segmentering jämfört med modeller tränade med konventionella, begränsade träningsdata. Detta bekräftar för forskarna att tillhandahållande av en mer varierad datauppsättning under träning av segmenteringsmodellen förbättrar prestandan.
Genom dessa förbättrade segmenteringsmöjligheter kommer forskarna att bättre kunna upptäcka celler och studera variabilitet mellan enskilda celler, särskilt bland stamceller. I framtiden hoppas forskarna kunna använda tekniken de har utvecklat för att gå bortom stillbilder för att generera videor, vilket kan hjälpa dem att lokalisera vilka faktorer som påverkar en cells öde tidigt i dess liv och förutsäga deras framtid.
"Vi genererar syntetiska bilder som också kan förvandlas till en time lapse-film, där vi kan generera den osedda framtiden för celler," sa Shariati. "Med det vill vi se om vi kan förutsäga framtida tillstånd för en cell, som om cellen kommer att växa, migrera, differentiera eller dela sig."
Mer information: Abolfazl Zargari et al, Enhanced Cell Segmentation with Limited Training Dataset using Cycle Generative Adversarial Networks, iScience (2024). DOI:10.1016/j.isci.2024.109740
Journalinformation: iScience
Tillhandahålls av University of California - Santa Cruz