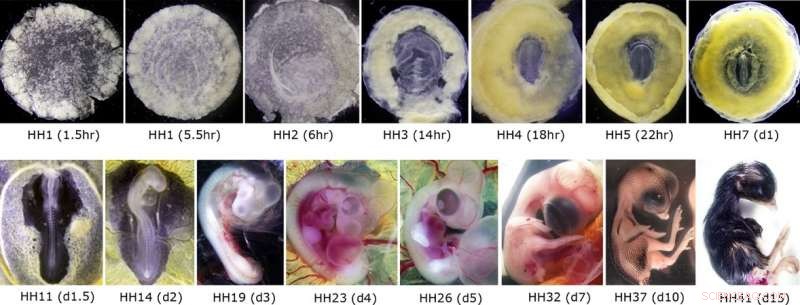
Representativa bilder av kycklingembryon som används för RNA-beredning och efterföljande CAGE-sekvensering. De övre panelerna visar embryon under den första dagen av embryonal utveckling (totalt 21 dagar), motsvarande de första fem veckorna av mänsklig graviditet (de första tre veckorna av mänsklig utveckling efter befruktning). Nedre paneler visar embryon från etableringen av cirkulationssystemet till ungefär en vecka före kläckning, liknande början av tredje trimestern i mänsklig utveckling. Kredit:Professor Guojun Sheng
Tidig utvecklingsforskning på mänskliga embryon är omöjlig på grund av etiska överväganden, och är tekniskt svår att utföra hos andra däggdjur, gör resurser för denna typ av forskning extremt sällsynta. Till exempel, projektet Functional Annotation of Mammalian Genome (FANTOM) 5 baserat i Japan, grundades 2000, är ett världsomspännande samarbetsprojekt med målet att identifiera alla funktionella element i däggdjursgenom, men den har ingen Cap Analysis of Gene Expression (CAGE)-baserad transkriptionsstartplats (TSS) data för mänskliga embryon eller för murina embryon som representerar tidiga utvecklingsstadier.
För att komma runt denna brist på information, ett internationellt samarbete mellan forskare från Japan (Kumamoto University &RIKEN), Ryssland (Kazan Federal University), Spanien (University of Cantabria), och Australien (University of Western Australia) har satt ihop världens första genomomfattande uppsättning av fågel-TSS. Dessa platser markerar startpunkten för transkription och är extremt viktiga för celldifferentiering och embryonutveckling fram till födseln, eller, när det gäller en fågelart, tills kläckning. Även om fåglar ser helt annorlunda ut än människor, tidiga utvecklingsstadier är mycket lika, och det har visat sig att cirka 60 % av mänskliga proteinkodande gener har en-till-en-överensstämmelse med kycklingortologer (gener som utvecklats från en gemensam förfader).
Genom att använda CAGE-teknik, en mycket pålitlig metod för att hitta TSS och cis-regulatoriska element (dessa sekvensblock som reglerar positionen och robustheten av transkription) i genomet under hela kycklingens utvecklingsperiod, kollaboratörerna kunde kartlägga 60 % av alla utvecklingsmässiga kyckling-TSS till det senaste kycklinggenomet, med de övriga 40% sannolikt ännu okarakteriserade alternativa promotorer eller icke-kodande RNA-gener. Det uppdaterade genomet lades till RIKENs interaktiva, onlineplattform ZENBU (det japanska ordet "zenbu" översätts till "alla" på engelska.) under namnet "Chicken-ZENBU" och är gratis för alla att använda.
Forskarna visade också att deras genomomfattande TSS-kartläggning kunde tillämpas på CRISPR-on-teknologi för att aktivera specifika gener under utveckling genom att framgångsrikt aktivera T-genen för Brachyury-protein som kodar i utvecklingsstadiet HH10 (ca 1,5 dagars utveckling efter läggning). Denna prestation följdes upp av flera mer framgångsrika genaktiveringar och visade därigenom effektiviteten av CRISPR-on i kombination med TSS-kartläggning.
"I processen att utveckla vår CAGE-baserade TSS-profileringsmetod för utveckling, vi identifierade nya transkriptionsfaktorer och deras regulatoriska moduler, samt flera nya hushållsgener, ", sade projektledaren professor Guojun Sheng vid Kumamoto Universitys IRCMS. "Vi förväntar oss att detta arbete och de data vi lagt till ZENBUs onlinedatabas kommer att avsevärt främja utvecklingsforskningen hos både fåglar och däggdjur."