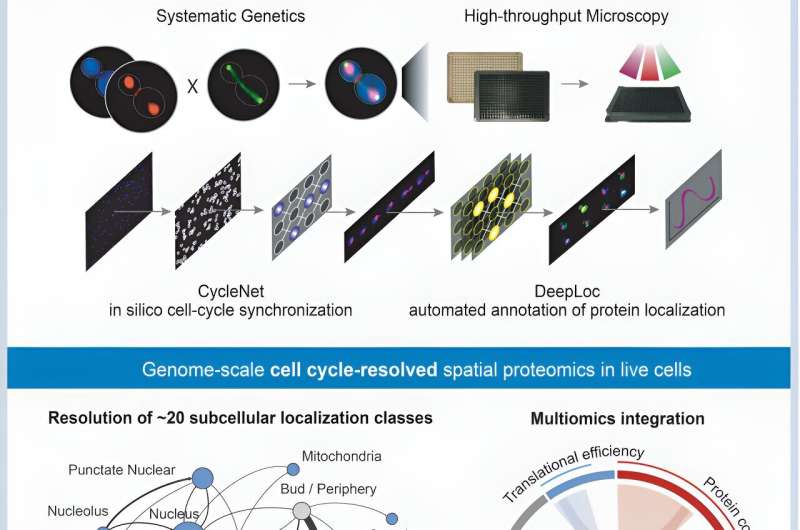
Ett internationellt team under ledning av forskare vid University of Toronto har kartlagt rörelsen av proteiner som kodas av jästgenomet under hela dess cellcykel. Detta är första gången som alla proteiner i en organism har spårats över cellcykeln, vilket krävde en kombination av djupinlärning och mikroskopi med hög genomströmning.
Teamet använde två konvolutionella neurala nätverk, eller algoritmer, kallade DeepLoc och CycleNet, för att analysera bilder av miljontals levande jästceller. Resultatet var en omfattande karta som identifierar var proteiner finns och hur de rör sig och förändras i överflöd i cellen under varje fas av cellcykeln.
"Vi fann att proteiner som regelbundet ökar och minskar i koncentrationen i cellen tenderar att vara involverade i att reglera cellcykeln, medan proteiner med förutsägbar rörelse genom cellen tenderar att underlätta cykelns biofysiska implementering", säger Athanasios Litsios, första författare till studien. och postdoktor vid U of T's Donnelly Center for Cellular and Biomolecular Research.
Studien publicerades i tidskriften Cell .
Cellcykeln förstås vara de stadier genom vilka en cell fortskrider för att slutligen dela sig i separata celler. Det är denna process som ligger till grund för spridningen av liv – och som pågår i allt levande.
På molekylär nivå beror cellcykeln på koordinationen av många proteiner för att överföra cellen från tillväxt och DNA-replikation hela vägen till celldelning. Dysregulation av proteiner kan kasta en skiftnyckel in i cellcykeln, med dess störning potentiellt leda till sjukdomar som cancer.
Forskarna observerade att omkring en fjärdedel av kartlagda jästproteiner följde regelbundna mönster av uppkomst och försvinnande eller förflyttning till specifika områden av cellen. De flesta proteiner följde dessa mönster för antingen koncentration eller rörelse, men inte båda.
"Vi identifierade cirka 400 proteiner med endast periodisk lokalisering under cellcykeln och cirka 800 med endast periodisk koncentration", säger Litsios. "Detta betyder att proteiner regleras på flera nivåer för att säkerställa att cellcykeln sker som programmerat."
Forskargruppen använde fluorescensmikroskopi för att spåra cirka 4 000 proteiner i bilder av jästceller för att klassificera cellcykelfasen samt platsen för proteiner inom 22 kategoriserade områden av cellen, såsom kärnan, cytoplasman och mitokondrierna. Identifiering av fas och proteinlokalisering automatiserades genom användning av konvolutionella neurala nätverk, med en cellcykelfasförutsägelse noggrannhet på mer än 93 %.
"Vi analyserade bilder av mer än 20 miljoner levande jästceller, som vi tilldelade olika cellcykelstadier med hjälp av maskininlärning", säger Brenda Andrews, huvudutredare för studien och universitetsprofessor i molekylär genetik vid Donnelly Center och Temerty Faculty of Medicin.
"Vi utvecklade och tillämpade sedan en andra beräkningspipeline för att undersöka hur proteiner förändras i lokalisering och koncentration under cellcykeln. Den här studien producerade en unik datauppsättning som erbjuder en genomskalig bild av molekylära förändringar som inträffar under celldelning."
"Jästcellen är en bra modell för eukaryotisk biologi", sa Litsios. "Det finns vissa saker vi kan göra med jästceller men inte med andra organismer som är antingen mer enkla eller komplexa. Vi kan använda jästceller för att observera processer i stor skala, vilket gör den till den perfekta organismen för att studera cellcykeln— i hopp om att bättre förstå den mänskliga cellcykeln."
Mer information: Athanasios Litsios et al, Proteom-skala rörelser och kompartment anslutning under den eukaryota cellcykeln, Cell (2024). DOI:10.1016/j.cell.2024.02.014
Journalinformation: Cell
Tillhandahålls av University of Toronto