Att förstå mutationsprocesser i en cell ger ledtrådar till utvecklingen av ett genom. Mest aktivt studeras mutationsprocesser i mänskliga cancerceller, medan andra genom ofta försummas.
En forskargrupp undersökte och jämförde det övergripande utbudet av mutationer i laboratorie- och naturliga E. coli (tarmbakterie), och kom fram till att de praktiskt taget inte går att särskilja. Resultaten presenteras i Genome Biology and Evolution journal.
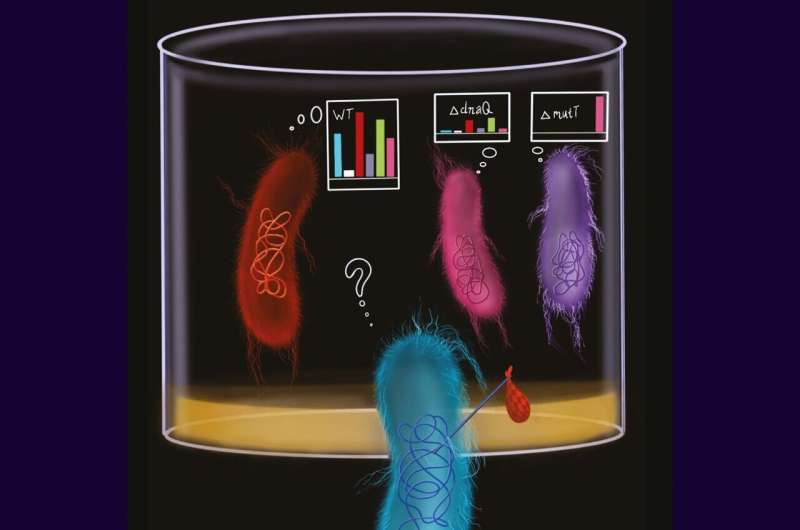
"Mutationer uppstår antingen i replikeringsprocessen – kopiering av DNA, eller när man "reparerar" DNA på grund av strängbrott eller andra störningar. Reparationssystemet kan också göra misstag.
"Olika replikerings- och reparationssystem gör olika misstag, och de beror på sammanhanget. Tre nukleotider undersöks vanligtvis:den där mutationen har inträffat och två på höger och vänster sida av den.
"En typisk uppsättning mutationer, tillsammans med de sammanhang som är specifika för ett visst system, kallas en mutationssignatur. Människor har till exempel mutationssignaturer av UV-exponering och rökning som är karakteristiska för vissa cancerformer", säger studiens chef. Professor Mikhail Gelfand, chef för Skoltechs Bio Center.
Teamet genomförde en första studie någonsin på mutationsprofilen för E. coli - summan av mutationssignaturer för replikering och reparationssystem. Det första steget var att testa uppgiften på gymnasieelever vid Summer School of Molecular and Theoretical Biology, där forskare också kom i kontakt med indiska kollegor som experimentellt arbetade med samma problem.
"Våra partners från Indien tillhandahöll data om E. coli med störda replikerings- och reparationssystem. Från dem kunde vi extrahera rena mutationssignaturer som är karakteristiska för olika system, och sedan dekomponerade mutationsprofilerna för E. coli till signaturer och förstod vilka system bidra till mutationer mer", fortsatte Gelfand.
Forskarna använde också experimentella data om den långsiktiga utvecklingen av E. coli. Denna labbstudie, som har pågått i över 30 år, leds av Richard Lenski. Mutationsprofilen för laboratoriestammar jämfördes med den för naturliga stammar.
"Vi fann att dessa profiler liknar varandra. Laboratoriestammen lever inte under förhållanden som liknar dem i naturen, så det här resultatet är oväntat. Dessutom talar resultaten emot tanken att utvecklingen av E. coli i det naturliga miljö inkluderar alternerande episoder av normal långsam evolution och kortare episoder av accelererad evolution i ett hypermuterbart läge.
"Tvärtom, under utvecklingen av naturliga stammar av E. coli, är bidraget från atypiska mutationsregimer till den totala ackumuleringen av mutationer obetydligt," sa Gelfand.
Mer information: Sofya K Garushyants et al, Mutational Signatures in Wild Type Escherichia coli-stammar avslöjar dominans av DNA-polymerasfel, Genome Biology and Evolution (2024). DOI:10.1093/gbe/evae035
Journalinformation: Genombiologi och evolution
Tillhandahålls av Skolkovo Institute of Science and Technology