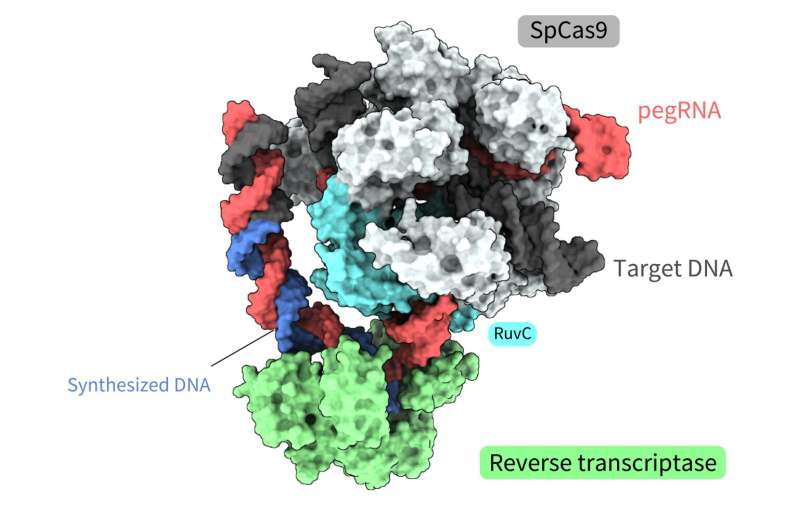
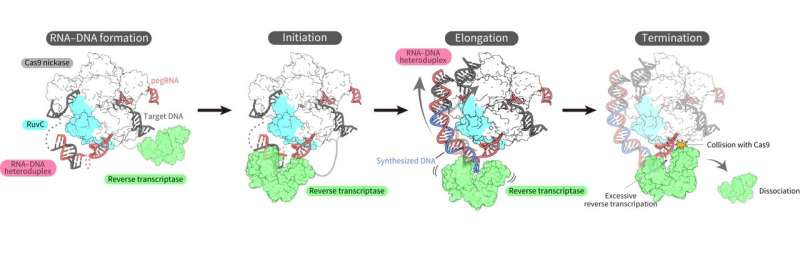
Gemensam forskning ledd av Yutaro Shuto, Ryoya Nakagawa och Osamu Nureki vid University of Tokyo bestämde den rumsliga strukturen för olika processer i ett nytt genredigeringsverktyg som kallas "prime editor". Funktionsanalys baserad på dessa strukturer avslöjade också hur en "prime editor" kunde uppnå omvänd transkription, syntetisera DNA från RNA, utan att "klippa" båda strängarna i dubbelhelixen.
Att klargöra dessa molekylära mekanismer bidrar i hög grad till att utforma genredigeringsverktyg som är tillräckligt noggranna för genterapibehandlingar. Resultaten publiceras i tidskriften Nature .
2020 års Nobelpris i kemi tilldelades Jennifer Doudna och Emmanuelle Charpentier för att de utvecklat ett banbrytande men ändå enkelt sätt att redigera DNA, "blåkopian" av levande organismer. Medan deras upptäckt öppnade nya vägar för forskning, begränsade metodens noggrannhet och säkerhetsproblem när det gäller att "klippa" båda DNA-strängarna dess användning för genterapibehandlingar. Som sådan har forskning pågått för att utveckla verktyg som inte har dessa nackdelar.
Prime-redigeringssystemet är ett sådant verktyg, ett molekylkomplex som består av två komponenter. En komponent är huvudredigeraren, som kombinerar ett SpCas9-protein, som används i den första CRISPR-Cas-genredigeringsteknologin, och ett omvänt transkriptas, ett enzym som transkriberar RNA till DNA.
Den andra komponenten är det primära redigeringsguide-RNA (pegRNA), ett modifierat guide-RNA som identifierar målsekvensen i DNA:t och kodar för den önskade redigeringen. I detta komplex fungerar den primära editorn som en "ordbehandlare", som exakt ersätter genomisk information. Verktyget har redan framgångsrikt implementerats i levande celler hos organismer som växter, zebrafiskar och möss. Men exakt hur detta molekylkomplex utför varje steg i redigeringsprocessen har inte varit klart, mestadels på grund av brist på information om dess rumsliga struktur.
"Vi blev nyfikna på hur den onaturliga kombinationen av proteinerna Cas9 och omvänt transkriptas fungerar tillsammans", säger Shuto, uppsatsens första författare.
Forskargruppen använde kryogen elektronmikroskopi, en avbildningsteknik som gör observationer möjliga i nära atomär skala. Metoden krävde att proverna var i glasig is för att skydda dem från potentiell skada från elektronstrålarna, vilket innebar ytterligare utmaningar.
"Vi fann att prime editor-komplexet var instabilt under experimentella förhållanden", förklarar Shuto. "Så det var mycket utmanande att optimera förutsättningarna för att komplexet skulle hålla sig stabilt. Under lång tid kunde vi bara bestämma strukturen för Cas9."
För att äntligen övervinna utmaningarna, lyckades forskarna bestämma den tredimensionella strukturen av prime editor-komplexet i flera tillstånd under omvänd transkription på mål-DNA:t.
Strukturerna avslöjade att det omvända transkriptaset band till RNA-DNA-komplexet som bildades längs "delen" av Cas9-proteinet associerat med DNA-klyvning, splittringen av en enkelsträng av dubbelhelixen. Medan den omvända transkriptionen utfördes bibehöll det omvända transkriptaset sin position i förhållande till Cas9-proteinet. De strukturella och biokemiska analyserna visade också att det omvända transkriptaset kunde leda till ytterligare, oönskade insättningar.
Dessa rön har öppnat nya vägar för både grundforskning och tillämpad forskning. Så, Shuto lägger ut nästa steg.
"Vår strukturbestämningsstrategi i denna studie kan också tillämpas på prime editors som består av ett annat Cas9-protein och omvänt transkriptas. Vi vill använda den nyligen erhållna strukturella informationen för att leda till utvecklingen av förbättrade prime editors."
Mer information: Strukturell grund för pegRNA-styrd omvänd transkription av huvudredigeraren, Nature (2024). DOI:10.1038/s41586-024-07497-8 , https://www.nature.com/articles/s41586-024-07497-8
Journalinformation: Natur
Tillhandahålls av University of Tokyo